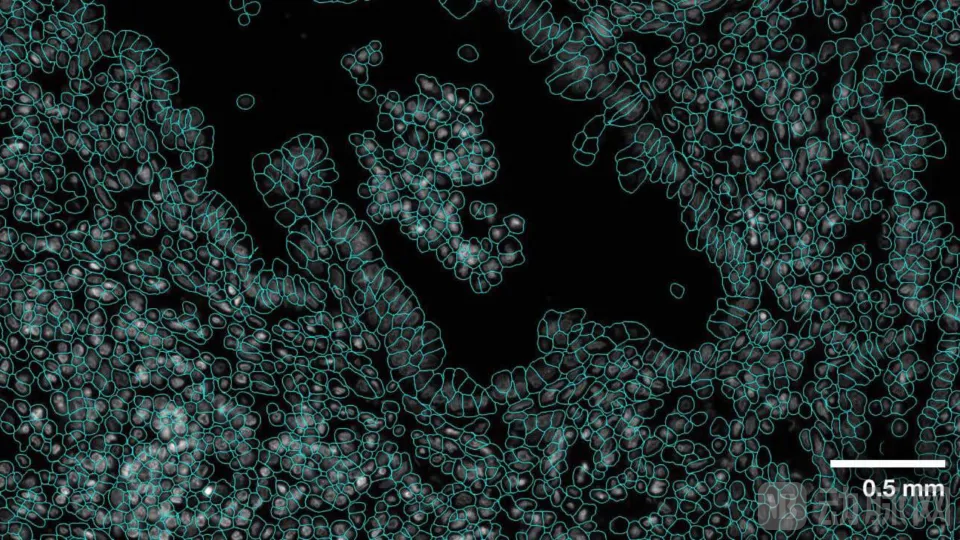
VISTA-2D 是 NVIDIA 推出的全新基础模型,能够快速、准确地执行细胞分割。细胞分割是一项细胞图像和空间组学工作流中的基本任务,对所有下游任务的准确性至关重要。
VISTA-2D 模型使用图像编码器创建图像嵌入,然后将其转换成分割掩码(图 1)。嵌入必须包含每个细胞的形态信息。
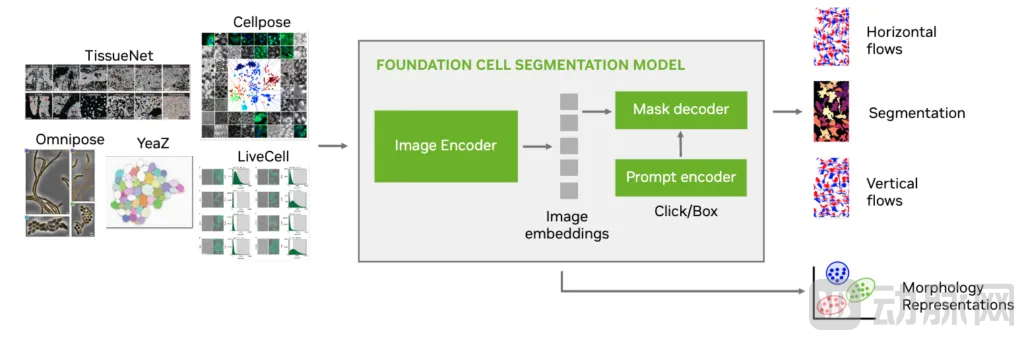
图 1. VISTA-2D 网络架构
如果能为每个细胞分割生成一个嵌入,那就可以对所有嵌入进行聚类,并自动将形态相似的细胞聚集在一起。
本文将深入介绍随附的 Jupyter Notebook,展示如何创建一个将细胞快速分类的自动流程。首先,通过这些工具,使用 VISTA-2D 对细胞进行分割并提取其空间特征,其次,使用 RAPIDS 对这些细胞特征进行聚类。
如要学习本教程,您需要:
基本了解 Python、Jupyter 和 Docker
有 Docker 版本 19.03+
本 Jupyter Notebook 的代码位于 /clara-parabricks-workflows/vista2d_rapids_clustering GitHub 资源库中,并在 NGC 的 PyTorch Docker 容器中运行。该 notebook 使用该容器的 24:03-py3 标签创建。
请使用以下命令运行该容器:

此命令启动以下操作:
启动 Docker 容器。
将该资源库的文件夹挂载到容器中。
将主机上的端口 8888 映射到 Docker 内的端口 8888。
为容器分配所有可用的 GPU。
启动 PyTorch 容器。
返回终端。
接下来,您需要一些其他 Python 软件包,这些软件包可以在 requirements.txt 中找到。

这些软件包主要用于辅助函数和绘图,本文将在后半部分进行进一步的说明。现在,可以将它们安装在 Docker 容器之上:

接下来,启动 notebook:

现在 notebook 服务器正在运行,可以在运行服务器的机器上或者在另一台机器上使用网络浏览器访问 notebook。
在浏览器中输入服务器所在机器的 IP 地址,然后输入端口 8888:

现在 notebook 已准备好运行。更多信息,请参见 GitHub 资源库。
该 notebook 的前半部分同时使用活体细胞数据与 VISTA-2D,来分割图像中的细胞,并使用 VISTA-2D 模型本身的编码层来提取特征。
首先,加载一个 VISTA-2D 模型检查点,这是因为该 notebook 的重点不是训练模型,而是使用模型进行特征提取。

接下来加载辅助函数,以避免主 notebook 过于冗长。

接下来的章节将详细介绍这些辅助函数的作用,它们都可以在 segmentation.py 中找到。
segment_cells
此函数获取细胞图像,并通过 VISTA-2D 自始至终进行处理。这将产生两张额外的图像:一张是完整的分割图像,另一张是每个细胞都标注了从 1 到图像中发现的细胞数(在 notebook 中称为 pred_mask)的图像。这使得细胞能够在后续进行特征提取时被单独索引。

plot_segmentation
该函数获取 segment_cells 的输出结果并显示图像,以便直观地验证分割和预测掩码的准确性。图 2 显示了使用 notebook 提供的细胞图像所输出的示例。

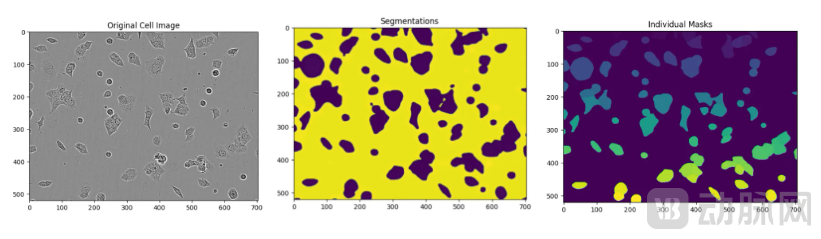
图 2. VISTA-2D 分割结果
通过三张图像显示 VISTA-2D 分割的结果:
原始细胞图像、从背景中分割出的所有细胞,以及每个细胞的单独掩码
feature_extract
该函数提取每个单独细胞的分割结果,并生成一个特征向量。每个细胞都包含在一个正方形掩码中。该掩码经过裁剪,大小只适合一个细胞和周围的背景。它使用 VISTA-2D 模型的前半部分作为编码器,来生成这些特征向量。
我们认为生成的向量包含细胞分割所需的所有信息,因此也必须包含每个细胞的形态信息。这些信息作为向量可以轻松输入到聚类算法中。形态相似的细胞应具有相似的特征向量,并被分配到相似的聚类中。

这样得到的矩阵行数为 num_cells,列数为 1024,即每个细胞的编码向量长度。
有了每个细胞的特征向量后,就可以使用 RAPIDS 的聚类算法运行它们了。
RAPIDS 是一个 GPU 提供加速的机器学习库,带有匹配常用 Python 数据科学库(如 pandas 和 sci-kit learn 等)的 API。虽然在该 notebook 中只需使用 RAPIDS 的特征缩减和聚类部分,但其实还有更多可用的功能。

TruncatedSVD
从 VISTA-2D 得到的特征向量长度为 1024。但由于图像中只有大约 80 个细胞,因此用这么多特征来建立聚类是没有意义的。
您可以使用降维算法来减少这些嵌入的长度,同时尽量减少丢失的信息。在该 notebook 中,可使用 Truncated SVD 算法将维数从 1024 维减少到 3 维。这样可以在 3D 空间中直观地看到聚类,更加方便绘制聚类图。
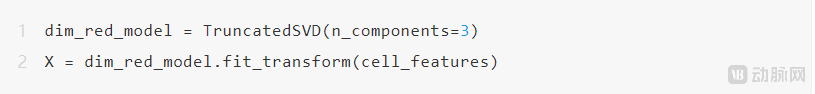
这样就得到了新的特征向量矩阵 X,其大小为 [num_cells, 3],而不是 cell_features 中原始向量的大小 [num_cells, 1024]。
DBSCAN
RAPIDS 中有很多聚类算法。在该 notebook 中,我们选择 DBSCAN。在这里,需要将 eps(两点之间的最大距离)设为 0.003,并将允许构成一个聚类的最小样本数设为 2。
现在运行 fit_predict 会为图像中的每个细胞生成一个聚类标签。如果将标签列表转换为标签字典,就可以更容易地查看哪些细胞被分配到了哪个群组。

最后,可以使用 Plotly 设置 3D 交互式绘图,显示每个细胞的聚类位置。

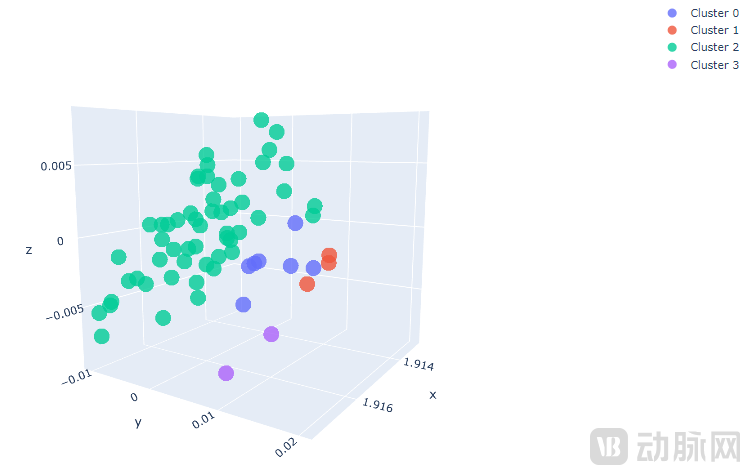
图 3. 根据聚类特征向量绘制的交互式 3D 图
本文向大家展示了如何使用 VISTA-2D 模型分割图像中的细胞,并从每个分割的细胞中提取特征向量,还演示了如何使用 RAPIDS 对这些向量进行聚类。
如要了解更多信息,请访问以下链接:
GitHub 上的 /vista2d 快速聚类 Jupyter Notebook:
https://github.com/clara-parabricks-workflows/vista2d_rapids_clustering
使用 NVIDIA AI Foundation 模型 VISTA-23 帮助进行细胞分割和形态分析:
https://developer.nvidia.com/blog/advancing-cell-segmentation-and-morphology-analysis-with-nvidia-ai-foundation-model-vista-2d/
RAPIDS 文档中提供降维和聚类信息的 API 参考文件:
https://docs.rapids.ai/api/cuml/stable/api/





























